Ricerca
Aggiornamento sullo stato di avanzamento: Il nostro ultimo modello AlphaFold mostra una precisione notevolmente migliorata ed espande la copertura oltre le proteine ad altre molecole biologiche, compresi i ligandi.
Dalla sua uscita nel 2020, AlphaFold ha rivoluzionato il modo in cui vengono comprese le proteine e le loro interazioni. Google DeepMind e Laboratori isomorfi hanno lavorato insieme per costruire le basi di un modello di intelligenza artificiale più potente che espanda la copertura oltre le sole proteine all’intera gamma di molecole biologicamente rilevanti.
Oggi lo siamo condividendo un aggiornamento sui progressi verso la prossima generazione di AlphaFold. Il nostro ultimo modello ora può generare previsioni per quasi tutte le molecole del pianeta Banca dati sulle proteine (PDB), raggiungendo spesso la precisione atomica.
Sblocca nuove conoscenze e migliora significativamente la precisione in più classi chiave di biomolecole, inclusi ligandi (piccole molecole), proteine, acidi nucleici (DNA e RNA) e quelli contenenti modifiche post-traduzionali (PTM). Questi diversi tipi di strutture e complessi sono essenziali per comprendere i meccanismi biologici all’interno della cellula e sono difficili da prevedere con elevata precisione.
Le capacità e le prestazioni ampliate del modello possono aiutare ad accelerare le scoperte biomediche e a realizzare la prossima era della “biologia digitale”, fornendo nuove informazioni sul funzionamento dei percorsi delle malattie, sulla genomica, sui materiali biorinnovabili, sull’immunità delle piante, sui potenziali bersagli terapeutici, sui meccanismi per la progettazione dei farmaci e nuove piattaforme per consentire l’ingegneria proteica e la biologia sintetica.
Serie di strutture previste rispetto alla verità fondamentale (bianco) dal nostro ultimo modello AlphaFold.
Al di là del ripiegamento delle proteine
AlphaFold è stata una svolta fondamentale per la previsione delle proteine a catena singola. Multimetro AlphaFold poi espanso a complessi con catene proteiche multiple, seguito da AlphaFold2.3, che ha migliorato le prestazioni e ampliato la copertura a complessi più grandi.
Nel 2022, le previsioni sulla struttura di AlphaFold per quasi tutte le proteine catalogate conosciute dalla scienza sono stati resi disponibili gratuitamente tramite il Database della struttura delle proteine AlphaFoldin collaborazione con l’Istituto europeo di bioinformatica dell’EMBL (EMBL-EBI).
Ad oggi, 1,4 milioni di utenti in oltre 190 paesi hanno avuto accesso al database AlphaFold e gli scienziati di tutto il mondo hanno utilizzato le previsioni di AlphaFold per contribuire a far avanzare la ricerca su tutto, dall’accelerazione di nuove tecnologie vaccini contro la malaria e avanzando scoperta di farmaci antitumorali allo sviluppo enzimi mangia-plastica per contrastare l’inquinamento.
Qui mostriamo le straordinarie capacità di AlphaFold di prevedere strutture accurate oltre il ripiegamento delle proteine, generando previsioni di strutture altamente accurate attraverso ligandi, proteine, acidi nucleici e modifiche post-traduzionali.
Prestazioni attraverso complessi proteina-ligando (a), proteine (b), acidi nucleici (c) e modifiche covalenti (d).
Accelerare la scoperta di farmaci
Le prime analisi mostrano anche che il nostro modello supera di gran lunga AlphaFold2.3 su alcuni problemi di previsione della struttura proteica che sono rilevanti per la scoperta di farmaci, come il legame degli anticorpi. Inoltre, prevedere con precisione le strutture dei ligandi proteici è uno strumento incredibilmente prezioso per la scoperta di farmaci, poiché può aiutare gli scienziati a identificare e progettare nuove molecole, che potrebbero diventare farmaci.
L’attuale standard industriale prevede l’utilizzo di “metodi di docking” per determinare le interazioni tra ligandi e proteine. Questi metodi di docking richiedono una struttura proteica di riferimento rigida e una posizione suggerita a cui legarsi il ligando.
Il nostro ultimo modello stabilisce un nuovo standard per la previsione della struttura proteina-ligando superando i migliori metodi di docking riportati, senza richiedere una struttura proteica di riferimento o la posizione della tasca del ligando, consentendo previsioni per proteine completamente nuove che non sono state strutturalmente caratterizzate prima.
Può anche modellare congiuntamente le posizioni di tutti gli atomi, consentendogli di rappresentare la piena flessibilità intrinseca delle proteine e degli acidi nucleici mentre interagiscono con altre molecole, cosa non possibile utilizzando i metodi di docking.
Ecco, ad esempio, tre casi recentemente pubblicati e rilevanti dal punto di vista terapeutico in cui le strutture previste dal nostro ultimo modello (mostrate a colori) corrispondono strettamente alle strutture determinate sperimentalmente (mostrate in grigio):
- PORCN: Una molecola antitumorale in stadio clinico legata al suo bersaglio, insieme a un’altra proteina.
- KRAS: Complesso ternario con un ligando covalente (una colla molecolare) di un importante bersaglio del cancro.
- PI5P4Kγ: Inibitore allosterico selettivo di una lipide chinasi, con molteplici implicazioni patologiche tra cui cancro e disturbi immunologici.
Previsioni per PORCN (1), KRAS (2) e PI5P4Kγ (3).
Isomorphic Labs sta applicando questo modello AlphaFold di nuova generazione alla progettazione di farmaci terapeutici, contribuendo a caratterizzare in modo rapido e accurato molti tipi di strutture macromolecolari importanti per il trattamento delle malattie.
Nuova comprensione della biologia
Sbloccando la modellazione delle strutture di proteine e ligandi insieme agli acidi nucleici e a quelli contenenti modifiche post-traduzionali, il nostro modello fornisce uno strumento più rapido e accurato per esaminare la biologia fondamentale.
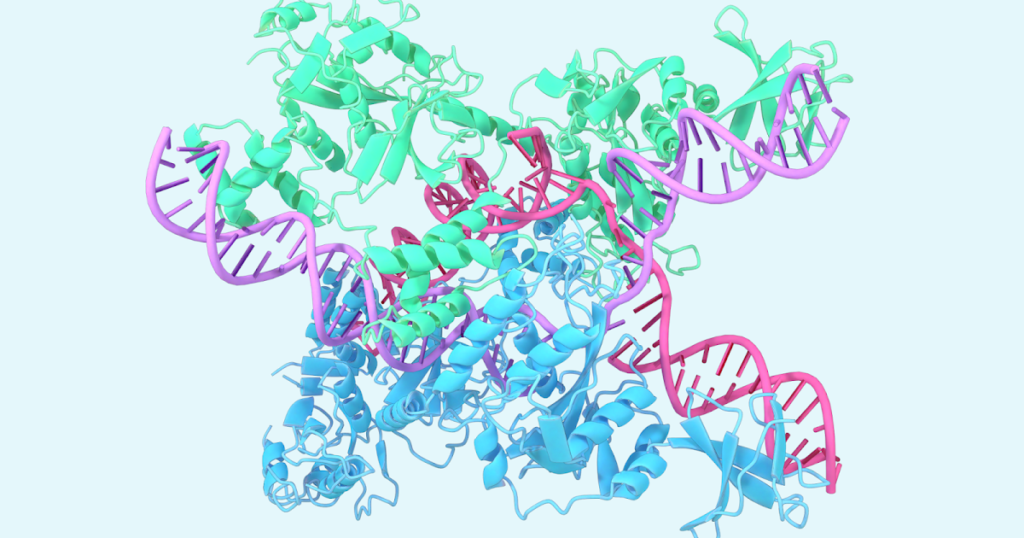
Un esempio riguarda la struttura di CasLambda legato al crRNA e al DNAparte di Famiglia CRISPR. CasLambda condivide la capacità di modifica del genoma di Sistema CRISPR-Cas9comunemente note come “forbici genetiche”, che i ricercatori possono utilizzare per modificare il DNA di animali, piante e microrganismi. Le dimensioni più piccole di CasLambda potrebbero consentire un uso più efficiente nell’editing del genoma.
Struttura prevista di CasLambda (Cas12l) legata al crRNA e al DNA, parte del sottosistema CRISPR.
L’ultima versione della capacità di AlphaFold di modellare sistemi così complessi ci mostra che l’intelligenza artificiale può aiutarci a comprendere meglio questi tipi di meccanismi e ad accelerarne l’uso per applicazioni terapeutiche. Altri esempi sono disponibile nel nostro aggiornamento sui progressi.
Avanzare l’esplorazione scientifica
Il drammatico salto di prestazioni del nostro modello mostra il potenziale dell’intelligenza artificiale nel migliorare notevolmente la comprensione scientifica delle macchine molecolari che compongono il corpo umano e del più ampio mondo della natura.
AlphaFold ha già catalizzato importanti progressi scientifici in tutto il mondo. Ora, la prossima generazione di AlphaFold ha il potenziale per contribuire a far avanzare l’esplorazione scientifica alla velocità digitale.
I nostri team dedicati di Google DeepMind e Isomorphic Labs hanno fatto grandi passi avanti in questo lavoro fondamentale e non vediamo l’ora di condividere i nostri continui progressi.
Fonte: deepmind.google


